GluonでDeep Learning:Tutorialを眺めてみる
導入
2017年10月12日(現地時間)に、MicrosoftとAWSがGluonというDeep Learningのライブラリを公開しました。
www.itmedia.co.jp
Gluonとは、自然界の基本的な相互作用の一つ「強い相互作用」を伝える素粒子のことです。glue(のり)にちなんでのりのように強く粒子を結びつけるといった感覚で名付けられました。ちなみに、ゲージ群でいうとSU(3)の随伴表現にアサインされます()。
立ち位置としては、TheanoやTensorflowに対するKerasに近い印象です。AWSがサポートしてるDeep LearningのフレームワークであるMXNetをサポートしており、コーディングを単純化・簡単化するようなものとなっています。Kerasと異なる点は、MXNetの一部のようになっている点でしょうか。Gluonは、インポートするのはあくまでMXNetで、その中のライブラリとしてGluonがある、といった感じになっています(触ってみた印象です)。
現在、Github上でDocumentやTutorialが公開されています。
github.com
こちらを見れば雰囲気はわかると思いますが、日本での紹介がまだ見当たらなかったので、眺めるついでに紹介をしたいと思います。
インストール方法
anacondaなどでPythonを導入していると、pipですぐインストールできます。ただし、インストールするのはmxnetです。
$ pip install mxnet
おそらく問題なくインストールできると思います。
Tutorial:MNIST
Githubのページには、MNISTで多層パーセプトロン(MLP)の実装例が載っています。
github.com
以下、Tutorialを眺めていきます。
ライブラリのインポート
mxnet、および必要なライブラリをインポートします。Gluonに対応するライブラリはgluonです。numpyは、Deep Learningのフレームワークに入力するデータの型をnp.XX32にする、とかいったデータの扱いまわりで必要なので、一緒にインポートしています。
import mxnet as mx from mxnet import gluon, autograd, ndarray import numpy as np
使用データセット:MNIST
手書き数字の画像と数字のラベルがセットになっているオープンなデータセットであるMNISTを使います。手書き数字の画像は28×28ピクセルで、各ピクセルに0から255の値が入っている2次元配列です(要はグレースケール)。各ピクセルの値を最大1になるように正規化の処理をし、型もnp.float32とします。このような処理をしないと、フレームワークによっては学習がうまく進まなかったりエラーになったりします。また、後で処理しますが、今回はMLPを想定しているので、画像データは28×28=784次元のベクトルデータとします。
Tutorialでは、MNISTのデータをmxnetのサイトからダウンロードしてきています。
train_data = mx.gluon.data.DataLoader(mx.gluon.data.vision.MNIST(train=True, transform=lambda data, label: (data.astype(np.float32)/255, label)), batch_size=32, shuffle=True) test_data = mx.gluon.data.DataLoader(mx.gluon.data.vision.MNIST(train=False, transform=lambda data, label: (data.astype(np.float32)/255, label)), batch_size=32, shuffle=False)
mx.gluon.data.DataLoaderですが、ロードしてきた(or 読み込んだ)データに対して、ミニバッチに分割してくれるメソッドです。ミニバッチのサイズや、シャッフルしてミニバッチにデータをアサインするかなどを指定できます。
trainデータは60000万枚、testデータは10000枚の画像データになっています。
ネットワーク構築
この部分がGluonの売りの一つになると考えられます。Gluonでは、Sequential()というメソッドを定義し、そこに積み木のように層を足していく感覚でネットワークのモデルを構築できます。KerasやCognitive Toolkitと似ていますね。
# モデルの初期化:Sequentialメソッドをインスタンス化 net = gluon.nn.Sequential() # addメソッドで層を追加する with net.name_scope(): net.add(gluon.nn.Dense(128, activation="relu")) net.add(gluon.nn.Dense(64, activation="relu")) net.add(gluon.nn.Dense(10)) # 出力層
Decse()は全結合層を足すメソッドで、ここでは引数としてユニットの数と活性化関数を指定しています。1層目と2層目のユニットの数はそれぞれ128と64で、活性化関数は両方ともReLUです。出力層に関しては、0~9の10種類の数字のクラス分類のタスクを考えるので、出力層の数は10とします。
出力層に関してコメントですが、活性化関数は学習の際に損失関数とセットで指定する思想のようです。ここはCognitive Toolkitと似ています。この思想のキモチは、モデルは「学習されるパラメータがある部分」ということなんだろうと個人的には思っています。クラス分類をする際のソフトマックス層は学習するパラメータはないので、ネットワークの出力を目的関数へ変換する際の一部分という位置づけになっているのだと思います。
学習の設定
ここはかなりCognitive Toolkit的です。パラメータの初期化、損失関数、最適化手法などを決め、それをTrainerというメソッド(どういう学習をするかのスケジューリングを司るもの)に放り込みます。
# パラメータ初期化:標準偏差0.05の正規乱数を利用 net.collect_params().initialize(mx.init.Normal(sigma=0.05)) # 損失関数はクロスエントロピー softmax_cross_entropy = gluon.loss.SoftmaxCrossEntropyLoss() # 最適化手法はSGDで、学習率は0.1 trainer = gluon.Trainer(net.collect_params(), 'sgd', {'learning_rate': .1})
これでネットワークのパラメータを学習する準備ができました。
学習
先程までの設定に従って学習していきます。何回データ学習に使うかの回数であるepoch数は10にしています。前にコメントしましたが、入力はミニバッチごとに投入、入力の形式はreshapeにより1次元のベクトルに変換しています。誤差逆伝搬に関係する微分操作などは、autogradが司っています。
# エポック数は10 epochs = 10 for e in range(epochs): for i, (data, label) in enumerate(train_data): data = data.as_in_context(mx.cpu()).reshape((-1, 784)) label = label.as_in_context(mx.cpu()) with autograd.record(): # 誤差逆伝搬をやる output = net(data) loss = softmax_cross_entropy(output, label) loss.backward() trainer.step(data.shape[0]) curr_loss = ndarray.mean(loss).asscalar() print("Epoch {}. Current Loss: {}.".format(e, curr_loss)) # 出力 Epoch 0. Current Loss: 0.08900555968284607. Epoch 1. Current Loss: 0.2502848505973816. Epoch 2. Current Loss: 0.2667727470397949. Epoch 3. Current Loss: 0.017404090613126755. Epoch 4. Current Loss: 0.006391774397343397. Epoch 5. Current Loss: 0.07984966039657593. Epoch 6. Current Loss: 0.009250413626432419. Epoch 7. Current Loss: 0.062348250299692154. Epoch 8. Current Loss: 0.06544484943151474. Epoch 9. Current Loss: 0.0023109368048608303.
実行すると、上のような感じでもりもり損失関数が小さくなっていきます。
その他、Gluonでできそうなこと
以上がTutorialになります。Tutorialだけだと予測も予実比較もしていないので、ネットワークの性能が知りたい場合は適宜追加で書く必要があります。
Documentを見る限りだと、MLP以外にもネットワークのモデルはサポートしているようです(今の時代当たり前だと思いますが)。ざっと見てみると
- 畳み込みニューラルネットワーク(CNN)
- 1, 2, 3次元の畳み込み層
- プーリング(Max, Avg, GlobalMax)
- 他テクニック
- BatchNormalizationやDropout
- ActivationにLeakyReLUがある
- 再帰型ニューラルネットワーク(RNN)
- RNN
- LSTM
- GRU
- Cell
- RNNCell, GRUCell, RecurrentCell, SequentialRNNCell, BidirectionalCell, DropoutCell, ZoneoutCell, ResidualCell
など、代表的なネットワークは作れる状態のようです。
Tutorialの内容の通り、比較的簡単にDeep Learningを試せるようなライブラリとなっています。また、今はMXNetだけですが、今後はCognitive ToolkitやCaffe2もサポートする予定とのことです。というわけで、複数のフレームワークを横断して使いやすいものになることが期待されるため、これからDeep Learningをやりたいと考えている人は検討するとよいのかもしれません。
Pythonでデータ分析:PyStanで線形回帰モデル
導入
ベイズ推定を行うための道具として、マルコフ連鎖モンテカルロ(MCMC)があります。その派生系であるハミルトニアンモンテカルロ(HMC)をベースにしたソフトウェアとして、Stanというものがよく知られています。
StanはC++ベースのソフトウェアですが、RやPythonを介しても使用することができます。今回は、PythonのインターフェースであるPyStanを使ってベイズ推定を行ってみます。
動機
Stan(特にRStan)は新人時代(2年半くらい前)に触っていたのですが、PJで機械学習の手法を使うことが多くなりあまり使わなくなり…といった感じでしばらく触れていませんでした。最近は、Pythonをベースに手を動かして幅を広げてみようと思っており、その一環としてPyStanに触れるのは良いだろうと思ったのが動機です。というわけで、基本的なことのメモ程度の紹介になると思います。
参考記事
流れを掴んだり、コードを書くに当たって、以下の記事が参考になりました。
qiita.com
使用データセット:Boston
久しぶりにボストン近郊の住宅情報のデータを使用します。
import sklearn from sklearn.datasets import load_boston from sklearn.model_selection import train_test_split Boston = load_boston() X = Boston.data y = Boston.target # 学習用・検証用にデータを分割 X_train, X_test, y_train, y_test = train_test_split(X, y, test_size = 0.2)
Stan部分
Stanに渡す部分を以下のように書きます。
# データ(辞書型) dat = {'N': X_train.shape[0], 'M': X_train.shape[1], 'X': X_train, 'y': y_train} # Stanコード model = """ data { int<lower=0> N; int<lower=0>M; matrix[N, M] X; vector[N] y; } parameters { real beta_0; vector[M] beta; real<lower=0> sigma; } model { for (i in 1:N) y[i] ~ normal(beta_0 + dot_product(X[i] , beta), sigma); } """
モデルに与えるデータは辞書型に変更しておきます。
Stanのコードは大まかに以下の3つの部分に分かれています。
- data
- parameters
- 推定するパラメータを定義します。ここも説明変数が多いときは、vectorで係数を与えるとコードがすっきりします。
- model
コンパイル
モデルをコンパイルします。結果はstmというオブジェクトにもたせておきます。
%time stm = StanModel(model_code=model) # 計算時間 CPU times: user 1.11 s, sys: 64 ms, total: 1.18 s Wall time: 1min 21s
計算時間も一緒に表示させていましが、1分半くらいかかっています(意外とかかるな。。)。
MCMC
サンプリングを実行します。iterationの回数は5000, 初期のiterationは安定的でないことが多いので推定には使わないことが多いのですが、使わないstep(n_warmup)は1000、マルコフ連鎖の数は2(特に大きな理由はないが)としています。
n_itr = 5000 n_warmup = 1000 chains = 2 # サンプリングの実行 %time fit = stm.sampling(data=dat, iter=n_itr, chains=chains, n_jobs=-1, warmup=n_warmup, algorithm="NUTS", verbose=False) # 計算時間 CPU times: user 556 ms, sys: 142 ms, total: 697 ms Wall time: 18min 50s
推定に約19分。ちょっと時間かかりますが、ローカルPCだとこんな程度でしょうか。
推定結果
パラメータの推定結果は、fitを出力させると表示できます。
fit * 出力結果 nference for Stan model: anon_model_0456a22b1cde927056973d34c99a2678. 2 chains, each with iter=5000; warmup=1000; thin=1; post-warmup draws per chain=4000, total post-warmup draws=8000. mean se_mean sd 2.5% 25% 50% 75% 97.5% n_eff Rhat beta_0 36.89 0.09 5.55 26.09 33.12 36.87 40.63 48.0 4151 1.0 beta[0] -0.11 3.9e-4 0.03 -0.18 -0.14 -0.11 -0.09 -0.05 8000 1.0 beta[1] 0.05 1.8e-4 0.01 0.02 0.04 0.05 0.06 0.08 6937 1.0 beta[2] 0.07 7.4e-4 0.07 -0.06 0.02 0.07 0.11 0.19 8000 1.0 beta[3] 2.52 0.01 1.0 0.57 1.84 2.52 3.19 4.49 8000 1.0 beta[4] -17.87 0.06 4.18 -25.99 -20.75 -17.78 -15.09 -9.63 5632 1.0 beta[5] 3.82 6.6e-3 0.47 2.91 3.5 3.83 4.14 4.74 5064 1.0 beta[6] -2.4e-3 1.6e-4 0.01 -0.03 -0.01-2.4e-3 7.7e-3 0.03 8000 1.0 beta[7] -1.34 2.7e-3 0.22 -1.76 -1.49 -1.35 -1.2 -0.91 6336 1.0 beta[8] 0.32 9.6e-4 0.07 0.17 0.27 0.32 0.37 0.46 5900 1.0 beta[9] -0.01 5.4e-5 4.2e-3 -0.02 -0.02 -0.01 -0.01-5.7e-3 6138 1.0 beta[10] -0.98 2.0e-3 0.14 -1.26 -1.08 -0.98 -0.89 -0.7 5230 1.0 beta[11] 8.6e-3 3.2e-5 2.9e-3 2.9e-3 6.7e-3 8.6e-3 0.01 0.01 8000 1.0 beta[12] -0.51 7.1e-4 0.06 -0.62 -0.55 -0.51 -0.47 -0.4 6223 1.0 sigma 4.61 1.9e-3 0.17 4.3 4.49 4.61 4.72 4.95 8000 1.0 lp__ -817.4 0.05 2.77 -823.7 -819.0 -817.1 -815.4 -812.9 3431 1.0 Samples were drawn using NUTS at Sat Oct 14 13:21:41 2017. For each parameter, n_eff is a crude measure of effective sample size, and Rhat is the potential scale reduction factor on split chains (at convergence, Rhat=1).
各パラメーターごとに平均、標準誤差、標準偏差、各パーセンタイル等を見ることができます。尤度に関する情報(lp__)もリストの一番下にあります。サンプリング結果の収束を判定する値として、Rhatというものがあり、Rhatが1.1以下だと十分収束していると判断できるようです。今回はすべての係数がRhat1.0となっているので、収束していると判断します。
推定結果の可視化
大雑把にはPyStanのplotメソッドで可視化できます。
fit.plot()
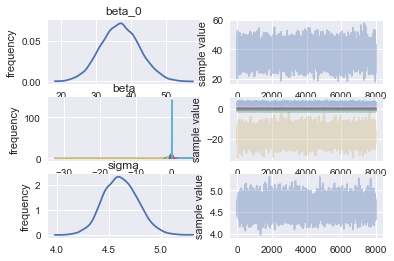
説明変数を行列で与えたのが災いして、係数の分布が見づらくなっています。そのため、もう少しわかりやすく可視化します。
# サンプル列を抽出 la = fit.extract(permuted=True) # パラメーター名 names = fit.model_pars # パラメーターの数 n_param = np.sum([1 if len(x) == 0 else x[0] for x in fit.par_dims]) # プロット f, axes = plt.subplots(n_param, 2, figsize=(15, 4*n_param)) cnt = 0 for name in names: dat = la[name] if dat.ndim == 2: for j in range(dat.shape[1]): d = dat[:,j] sns.distplot(d, hist=False, ax=axes[cnt, 0]) sns.tsplot(d, alpha=0.8, lw=.5, ax=axes[cnt, 1]) cnt += 1 else: # Intercept sns.distplot(dat, hist=False, ax=axes[cnt, 0]) sns.tsplot(dat, alpha=0.8, lw=.5, ax=axes[cnt, 1]) cnt += 1 name_list = [] for name in names: dat = la[name] if la[name].ndim == 2: for i in range(dat.shape[1]): name_list.append("{}{}".format(name,i+1)) else: name_list.append(name) for i in range(2): for j, t in enumerate(name_list): axes[j, i].set_title(t) plt.show()
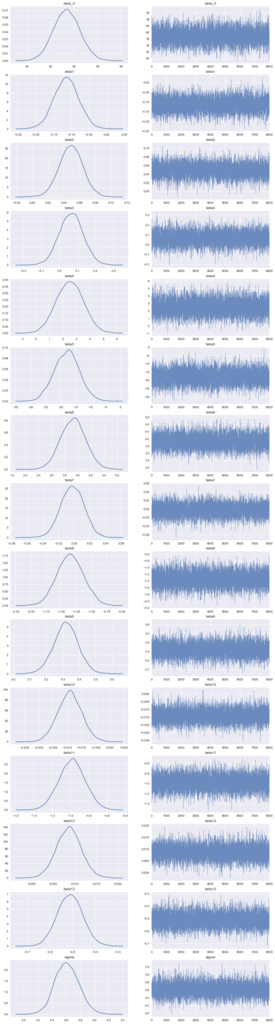
まあ概ね推定できているでしょう。分布的に幾つかは効果がほぼない変数もあるようです。精度や解釈性を向上させたい場合は、そのような変数はモデルから削除することもありますが、ここではこのまま進めます。
精度評価
得られた係数を用いて学習用・検証用データに対して予実比較を行います。今回は、簡単に係数の分布の平均値を推定された係数の値として予測値算出をします。
# 平均値算出 beta0 = la['beta_0'].mean() beta = la['beta'].mean(axis = 0) # 予測値算出 y_pred_train = (X_train * beta).sum(axis = 1) + beta0 y_pred_test = (X_test * beta).sum(axis = 1) + beta0 # 学習用、検証用データに関してMSEとR^2を出力 from sklearn.metrics import mean_squared_error, r2_score print('MSE Train: %.2f, MSE Test: %.2f' % (mean_squared_error(y_train, y_pred_train), mean_squared_error(y_test, y_pred_test))) print('R^2 Train: %.2f, R^2 Test: %.2f' % (r2_score(y_train, y_pred_train), r2_score(y_test, y_pred_test))) # 出力結果 MSE Train: 20.41, MSE Test: 28.30 R^2 Train: 0.75, R^2 Test: 0.70
以前自分が書いたsklearnを使った線形回帰モデルの記事では、R^2 Train : 0.763, Test : 0.659となっていました。データの分割の仕方に依存しそうですが、検証用に関しては若干R^2が改善しています。
tekenuko.hatenablog.com
Pythonでデータ分析:Catboost
導入
2017年7月に、ロシアのGoogleと言われている(らしい)Yandex社から、Catboostと呼ばれるGradient Boostingの機械学習ライブラリが公開されています。
ここ何ヶ月か調整さんになっていて分析から遠ざかりがちになりやすくなっていたのですが、手を動かしたい欲求が我慢できなくなってきたので、いい機会だと思って触ってみることにしました。
使用データセット:Titanic
Titanic号の乗客の情報および生存・死亡のデータを用いて、どの程度の精度のモデルができるかを試してみます。
データセットは、Kaggleのページからダウンロードしました。
Titanic: Machine Learning from Disaster | Kaggle
train.csvとtest.csvの2つを利用します。train.csvはSurvived(生存なら1、死亡なら0)のカラムがあるデータ、test.csvはSurvivedのカラムが無いデータになっています。train.csvを用いてモデルを作成し、test.csvからSurvivedの予測値を出し、Kaggleにsubmitすることで、モデルの精度(今回はAccuracy)を見ます。
train.csvのデータはdf_raw、test.csvのデータはdf_raw_testという名前で読み込んでおきます。
データ加工(笑)
Titanicのデータは、いくつか欠損があるカラムがあるのですが、今回は雑に-999で埋めます。また、乗客の名前などの非構造データも幾つかあるのですが、それらは予めインデックスを保持しておき、モデリングの際によしなにカテゴリカル変数として処理してもらうことにします(Catboostはそういった機能があったので、挙動確認も込めてあえて特徴量加工を頑張らないことにしました)。
# pandasやnumpyはインポート済み # 欠損値補完 df_raw.fillna(-999, inplace=True) # 説明変数・目的変数 X = df_raw.drop('Survived', axis = 1) y = df_raw['Survived'] # カテゴリカル変数として扱うカラム指定 categorical_features_indices = np.where(X.dtypes != np.float)[0]
訓練用と検証(Validation)用にデータを分割しておきます。
rom sklearn.model_selection import train_test_split X_train, X_validation, y_train, y_validation = train_test_split(X, y, test_size = 0.3)
モデリング
今回はクラス分類なので、catboost.CatBoostClassifierという関数を利用します。
import catboost mod = catboost.CatBoostClassifier(iterations=5000, calc_feature_importance = True, use_best_model=True, eval_metric = 'Accuracy' ) mod.fit(X_train, y_train, cat_features=categorical_features_indices, eval_set=(X_validation, y_validation), plot=True)
オプションについては、例えば以下のページにあります。
tech.yandex.com
今回は、CatBoostClassifierにはiterations:何回学習と検証を繰り返すか、calc_feature_importance:変数重要度を計算するか、use_best_model:iterationの中で一番検証用の指標が良いものを選ぶか、eval_metric:評価指標をオプションとして選んでいます。
モデル作成は、sklearnのようにfitメソッドで行います。cat_featuresでカテゴリカル変数として扱うカラムを指定し、eval_setで最初のvalidationに使用するデータを指定します。
実行後は、以下のような図がプロットされます。
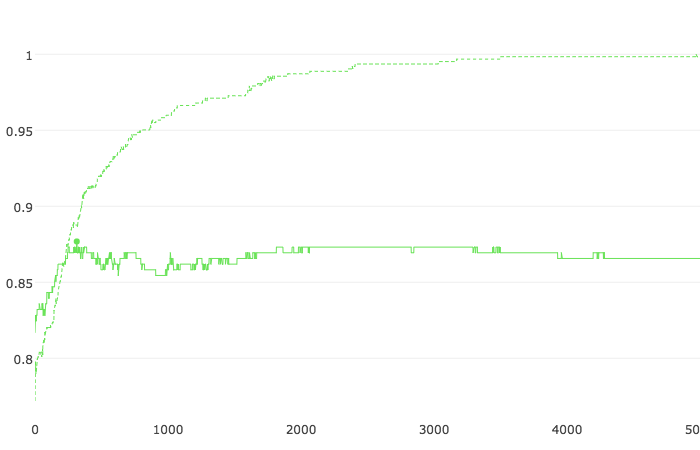
これは、訓練・検証用データに対してAUCをプロットしたものです。点線が訓練用、普通の線が検証用なので、結構過学習しちゃってます(汗)。検証用データで一番良い場合はaccuracyが0.88程度のようです。
from sklearn.metrics import accuracy_score y_val_pred = mod.predict(X_validation) print('accuracy : %.2f' % accuracy_score(y_val_pred, y_validation)) # 出力 accuracy : 0.88
テストデータを用いて、予測してみます。これはsklearnの場合と同様に、predictメソッドで出力できます。ただし、floatとして出て来るので、出力後にintに変換します。こうしておかないと、Kaggleにsubmitしたときにscoreが0になります。
# テストデータはdf_raw_test df_raw_test.fillna(-999, inplace = True) y_test = mod.predict(df_raw_test) est = DataFrame() test['PassengerId'] = df_raw_test['PassengerId'] test['Survived'] = y_test # 予測値をintに変換 test['Survived'] = test['Survived'].astype('int') # 保存 test.to_csv('out/benchmark_catboost.csv', index = False)
この結果をKaggleにsubmitすると....

ほぼほぼ何も頑張ってないのにaccuracyが0.8を超えました。ただ、過学習しているモデルなので、より安定して精度を出したかったらパラメータ調整などをおこなって汎化性能を上げる必要があるかと思います。そういった努力は今後の課題にします。
変数重要度
Catboostはfeature_importances_で変数の重要度も出力することができます(ソートはされていない)。ざっくり可視化しておきます。
FI = DataFrame() FI = FI.append(mod.feature_importances_) FI.index = X.columns FI.columns = ['Feature_Importance'] # 可視化 import matplotlib.pyplot as plt %matplotlib inline FI.plot(kind='bar')
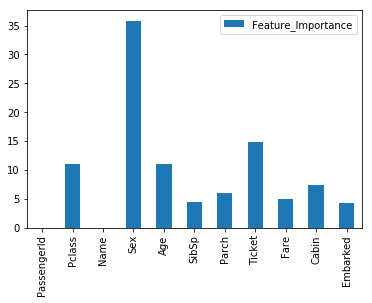
Pclassは乗客の社会的地位、Sexは性別、Ageは年代で、地位の高い女性や子供が優先的に救出されたという歴史的経緯が概ね反映されています。Ticletが効いているのはちょっと難しいですが。
Next Step
大雑把には使い方が分かったので、今後はGrid Searchなどを詰めていって、より使いこなせるようにしていこうと思います。
Memo:MacOS SierraでXGboostをpipで入れる
XGboostを自宅のMacに入れようとしても入らなかったので、調べてみたことを備忘録として残しておきます。
以前との差分を考えてみたら、MacOSをSierraにアップデートしてたことに気が付き、調べると以下の記事がヒットしました。
上の記事では、clang-ompのエイリアスの設定もやっているんですが、自分の場合はそれをやらなくてもXGboostがpipで入れられることを確認しました。またまたちょっと調べると、LLVM 8.1から公式のclangがOpenMPに対応したため、clang-ompはこれ以上更新されないと。そのため最新のLLVMを導入したほうがいいとのこと。自分はclang-ompを入れていないので、気にしなくても大丈夫(だったと理解)。
というわけで、OSが変わると色々面倒なことがあるということを再確認したのでした。
NN論文の読み会で発表した
はじめて外部勉強会なるもので発表をしました。
tfug-tokyo.connpass.com
今回の論文のテーマはDeep Learning + 自然言語処理系で、私は全然キャッチアップしてなかったところだったので、勉強(炎上ラーニング)を兼ねて申し込んでみました。私が選んだ論文は、Amazonが行っているソーシャルボット(対話型ボット)のコンペ出場チームの報告論文です。
www.slideshare.net
昔ながらの人工無脳から、NNを使った最近の手法をふんだんに使って複数の会話の応答候補を出力し、その中でよいものを深層強化学習(実際にはそこまで強化学習してない)で最適化するという内容で、いろいろ知識を得るのに非常に役立ちました。2週間くらいで前提知識ほぼゼロからキャッチアップして発表というスケジュールだったので、非常に苦しかったですが(笑)
発表はあまりうまくできなかった気がしますが、いろいろな人からたくさん質問をいただいて、非常に楽しい時間でした。また次回以降、もしくは別の勉強会でも発表して多くの人と交流していきたいと思いました。