Pythonでデータ分析:線形回帰モデル
導入
データ分析にて、最も基本的な回帰分析から始めていきます*1。回帰分析とは、説明したい変数(目的変数)とそれを説明するための変数(説明変数)の間の関係を求める手法です。機械学習の手法の区分としては、教師あり学習(解答に相当する教師データを用いてモデルを構築)に属するものです。
回帰分析の目的は大きく2種類あります。
- 理解:文字通り、変数間の構造・関係の理解
- 例:ある病気に対して、どのような遺伝子が寄与しているかをマイクロアレイデータなどのデータを用いて明らかにする
- 予測:変数間の関係をもとに、将来予測をする
- 例:過去の広告の投下量と売上実績から、これらの関係を説明するモデルを構築し、それをもとに将来の広告への投資計画を立てる
ビジネスの世界では、予測を目的に使用されることがほとんどです。それは、モデル構築によって要因を明らかにするだけでなく、その後の施策でそのモデルの知見を使い、価値を生み出すことが求められるからです。逆に、アカデミックでは理解が目的の研究も一定数あります。実務でデータ分析をやっている方は、例えば、マーケティングサイエンス学会に参加するとこのあたりの思想の違いを感じ取れて面白いと思います。
使用するデータセット
ボストン近郊の住宅情報のデータを使用します。こちらは、無償で提供されており、例えばUCI Machine Learning Repository(UCI Machine Learning Repository: Housing Data Set)からダウンロードすることが可能です。ここでは、scikit-learnからロードする形で導入します。
## 必要なライブラリのインポート from pandas import DataFrame from sklearn.datasets import load_boston ## データの用意 # ボストンデータをインポート boston = load_boston() # 説明変数たちをDataFrameへ変換 df = DataFrame(boston.data, columns = boston.feature_names) # 目的変数をDataFrameへ追加 df['MEDV'] = np.array(boston.target)
DataFrameという形式で保持するために、pandasを導入しています。最初の数行を表示してみます。
# 最初の5行を表示 df.head() # コンソールでの表示結果 >>> df.head() CRIM ZN INDUS CHAS NOX RM AGE DIS RAD TAX 0 0.00632 18.0 2.31 0.0 0.538 6.575 65.2 4.0900 1.0 296.0 1 0.02731 0.0 7.07 0.0 0.469 6.421 78.9 4.9671 2.0 242.0 2 0.02729 0.0 7.07 0.0 0.469 7.185 61.1 4.9671 2.0 242.0 3 0.03237 0.0 2.18 0.0 0.458 6.998 45.8 6.0622 3.0 222.0 4 0.06905 0.0 2.18 0.0 0.458 7.147 54.2 6.0622 3.0 222.0 PTRATIO B LSTAT MEDV 0 15.3 396.90 4.98 24.0 1 17.8 396.90 9.14 21.6 2 17.8 392.83 4.03 34.7 3 18.7 394.63 2.94 33.4 4 18.7 396.90 5.33 36.2
全部で14個のカラムがありますが、今回は最初の13個を説明変数、14個目を目的変数として使用することを想定しています。それぞれのカラムにある情報は以下です。
- CRIM : 犯罪発生率(人口単位)
- ZN : 25000平方フィート以上の住宅区間の割合
- INDUS : 非小売業の土地面積の割合(人口単位)
- CHAS : チャールズ川沿いかどうかフラグ(川沿いなら1、そうでなければ0)
- NOX : 窒素感化物の濃度(pphm単位)
- RM : 一戸あたりの平均部屋数
- AGE : 1940年よりも前に建てられた家屋の割合
- DIS : ボストンの主な5つの雇用圏までの重み付き距離
- RAD : 幹線道路へのアクセス指数
- TAX : 10000ドルあたりの所得税率
- PTRATIO : 教師あたりの生徒の数(人口単位)
- B : 1000(Bk - 0.63)^2として計算される量(Bkはアフリカ系アメリカ人居住者の割合(人口単位))
- LSTAT : 低所得者の割合
- MEDV : 住宅価格の中央値(単位は1000ドル)
線形回帰モデルの構築
通常はモデル構築に至るまでにデータ理解や基礎集計、データ加工といった非常に重要なステップがありますが、省略します。ここでは、目的変数をMEDV、説明変数をCRIM~LSTATとした回帰モデルを構築します。今回、目的変数は連続値とみなすことができるため、回帰モデルの中でも最も単純な、目的変数と説明変数間の関係が線形である場合を仮定します。
は回帰係数と呼ばれる量で、それぞれの変数がどれだけ目的変数に寄与するかを表しています。特に、
はすべての説明変数の寄与が0の場合も残る「ベース」的な量で、切片(Intercept)とも呼ばれます。
は残差と呼ばれる量で、各データとモデル式の「ズレ」を表します。線形回帰モデルでは、各データ点の残差の二乗を足し合わせたものを最小にするという条件のもとで、係数を求めます。
scikit-learnによる係数の推定
ここでは、いったん全データを用いて線形回帰モデルを構築(=回帰係数の推定)をします。scikit-learnにあるLinearRegressionを用いると、簡単に係数の推定が行えます。
## 説明変数と目的変数 # 説明変数 X = df.loc[:, boston.feature_names].values # 目的変数 y = df.loc[:, 'MEDV'].values ## 必要なライブラリのインポート from sklearn.linear_model import LinearRegression # オブジェクト生成 mod = LinearRegression(fit_intercept = True, normalize = False, copy_X = True, n_jobs = 1) # fit関数でパラメータ推定 mod.fit(X, y)
LinearRegressionの引数を明示的に書いてありますが、それぞれは以下の意味を持っています。
- fit_intercept : 切片項をモデル式に入れるかどうか。デフォルト値はTrue。
- normalize : 説明変数を正規化するかどうか。デフォルト値はFalse。
- copy_X : メモリ内でデータを複製してから実行するかどうか。 デフォルト値はTrue。
- n_jobs : 計算に使うジョブ数。デフォルト値は1で-1にするとすべてのCPUを使って計算。
回帰係数を出力してみます。
# 回帰係数を出力 print(mod.coef_) print(mod.intercept_) # 出力結果 >>>print(mod.coef_) [ -1.07170557e-01 4.63952195e-02 2.08602395e-02 2.68856140e+00 -1.77957587e+01 3.80475246e+00 7.51061703e-04 -1.47575880e+00 3.05655038e-01 -1.23293463e-02 -9.53463555e-01 9.39251272e-03 -5.25466633e-01] >>>print(mod.intercept_) 36.4911032804
学習データ、検証データに分割して性能評価
先程は、全データを用いて回帰係数の推定を行いました。しかし、それだけだと今持っているデータ「だけ」によく当てはまるモデルを作ってしまい、予測で良い性能を発揮しない場合があります。それを確認するための方法として、パラメータ推定の際に使用しなかったデータを用いて精度検証を行う、という方法があります。ここでは、持っているデータを学習用、検証用の2種類に分割して精度検証します。
上記のような精度検証は、scikit-learnを利用すると簡単に行うことができます。
# train_test_splitをインポート from sklearn.cross_validation import train_test_split # 70%を学習用、30%を検証用データにするよう分割 X_train, X_test, y_train, y_test = train_test_split(X, y, test_size = 0.3, random_state = 666) # 学習用データでパラメータ推定 mod.fit(X_train, y_train) # 作成したモデルから予測(学習用、検証用モデル使用) y_train_pred = mod.predict(X_train) y_test_pred = mod.predict(X_test)
残差プロット
モデルの性能を見る大雑把な方法として、残差プロットがあります。これは、予測値と実際の観測値の差分を可視化し、モデルで拾いきれていない非線形性や外れ値がないか、といった観点から、残差がランダムに分布しているかをチェックする方法です。数値的に性能を評価する前に、可視化して傾向を見ておくのは重要です。残差をプロットするコードは、例えば以下になります。
# matplotlibをインポート import matplotlib.pyplot as plt # Jupyterを利用していたら、以下のおまじないを書くとnotebook上に図が表示 %matplotlib inline # 学習用、検証用それぞれで残差をプロット plt.scatter(y_train_pred, y_train_pred - y_train, c = 'blue', marker = 'o', label = 'Train Data') plt.scatter(y_test_pred, y_test_pred - y_test, c = 'lightgreen', marker = 's', label = 'Test Data') plt.xlabel('Predicted Values') plt.ylabel('Residuals') # 凡例を左上に表示 plt.legend(loc = 'upper left') # y = 0に直線を引く plt.hlines(y = 0, xmin = -10, xmax = 50, lw = 2, color = 'red') plt.xlim([10, 50]) plt.show()
コードの実行結果は以下のようになります。
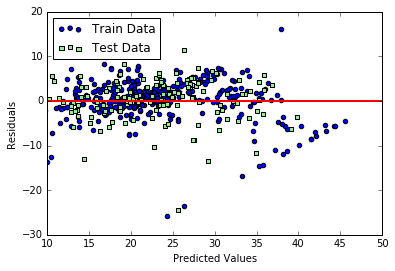
予測が完璧である場合は、残差はちょうど0になりますが、現実的にはそれはまずありません。良い回帰モデルの場合は、残差がちょうと0を中心にランダムに分布するような傾向が見えるはずです。今回のモデルに関しては、概ねランダムに分布しているようにも見えますが、学習用データを使った場合の残差に関してグラフの右下になにか直線的な傾向があり、モデルに取り切れていない情報がある可能性があります。
平均二乗誤差、決定係数
数値的にモデルの性能を評価する指標の例として、平均二乗誤差(Mean Squared Error : MSE)や決定係数()があります。これらは、データ点を
でラベル付けしたとすると、以下のように定義されるものです。
MSEは、誤差の二乗和の平均値で、学習用データ、検証用データともに小さければモデルの性能が良い、と判断されます。に関しては、MSEが0の場合に1をとる、つまりモデルの性能が良いほど1に近い量になります。また、
は学習用データに関しては0から1の値を取りますが、検証用データの場合には負の値を取ることもあります。
MSEとは、scikit-learnを用いると簡単に評価できます。これらを出力するコードは、以下のようになります。
# 平均二乗誤差を評価するためのメソッドを呼び出し from sklearn.metrics import mean_squared_error # 学習用、検証用データに関して平均二乗誤差を出力 print('MSE Train : %.3f, Test : %.3f' % (mean_squared_error(y_train, y_train_pred), mean_squared_error(y_test, y_test_pred))) # 学習用、検証用データに関してR^2を出力 print('R^2 Train : %.3f, Test : %.3f' % (mod.score(X_train, y_train), mod.score(X_test, y_test))) # 出力結果 >>>print('MSE Train : %.3f, Test : %.3f' % (mean_squared_error(y_train, y_train_pred), mean_squared_error(y_test, y_test_pred))) MSE Train : 21.371, Test : 24.336 >>>print('R^2 Train : %.3f, Test : %.3f' % (mod.score(X_train, y_train), mod.score(X_test, y_test))) R^2 Train : 0.763, Test : 0.659
ちなみに、はsklearn.metrics.r2_scoreを呼び出して評価することも可能です。結果を見ると、学習用データのほうが検証用データよりも
- MSEが小さい
が大きい
という傾向があり、過学習気味のモデルであることがわかります。
*1:これは、分析プロジェクトでいうモデル構築のフェーズでの基本という意味です。
Pythonでデータ分析:導入
目標
試してみたいデータがあった場合に、簡単な分析に関してはとっさに手が動くレベルをまず目指します。そのために、データ加工ではnumpyとpandas(あとseries)、モデル構築ではscikit-learn、可視化ではmatplotlibとseabornを交えて紹介していくことで、この目標を実現していこうと考えています*2。
進め方
データ分析をやる上でもっとも基本的と思われる回帰分析から始めていこうと思います。基本的なことがらではありますが
- 様々な拡張的手法のベースになっている
- ビジネスの世界では解釈のしやすさなどの理由で回帰分析がまだまだ大事
といった理由から、このアウトプットは無駄にはならないと期待しています。
線形回帰のあとは、回帰問題だけではなく、機械学習の手法を幾つか取り上げてまとめていきます。また、単なるライブラリの使用だけでなく、可能な範囲で実装もできたらよいなと考えています。
参考文献
以下の本では、ライブラリの使い方だけでなく、実装も交えて機械学習の手法が紹介されているので、こちらを参考にしていこうと考えています*3。
book.impress.co.jp
Rで粒子フィルタ:RcppSMCのテスト
経緯
Rで粒子フィルタ(逐次モンテカルロ)ってできるんだっけ、とふと思ったので少し調べてみました。すると、Rのパッケージでは、例えば
- https://cran.r-project.org/web/packages/SMC/SMC.pdf : {SMC}
- https://cran.r-project.org/web/packages/RcppSMC/RcppSMC.pdf : {RcppSMC}
というパッケージがあるようです。{RcppSMC}の方は、Rcppで開発されているパッケージです。まずは、簡単にテストできそうな{RcppSMC}を使ってみることにしました。
参考記事
ネットで探しているときに目についた方のブログを参考にしました。
というかほぼ丸パクリです。
d.hatena.ne.jp
ito-hi.blog.so-net.ne.jp
また、粒子フィルタに関する書籍も載せておきます。自分が読んだことあるものは、例えば以下です。ざっくり概要を知るのは樋口さんの本のほうがよさげです。
www.kspub.co.jp
時系列解析入門https://www.iwanami.co.jp/.BOOKS/00/4/0054550.htmlwww.iwanami.co.jp
テスト
パッケージのインストール
if(!require(RcppSMC)){ install.packages("RcppSMC", dependencies = TRUE) } require(RcppSMC)
テストデータの生成
線形ガウス状態空間モデルのシミュレーションデータを生成します。データ点は特になんの思想もなく100点にしました。
n <- 100 sim <- simGaussian(len = n)
simはlistで、状態変数(state)と、観測データ(data)が格納されています。
プロット
無駄に{ggplot2}で描きます。
# パッケージのインストール if(!require(ggplot2)){ install.packages("ggplot2", dependencies = TRUE) } require(ggplot2) # data.frameに変換 df <- data.frame(iteration = 1:length(sim$state), state = sim$state, data = sim$data) # プロット ggplot(data = df) + geom_line(mapping = aes(x = iteration, y = state), colour = "magenta") + geom_point(mapping = aes(x = iteration, y = data), shape = 21, size = 3, colour = "blue")

観測データから状態を推定
ここでは、逆に与えられた観測データから状態変数を推定します。{RcppSMC}では、blockpfGaussianOpt関数を使用して行います。plot = TRUEとすると、自動的に図を出力してくれます。ここで、粒子数は1000としています。
blockpfGaussianOpt(sim$data, particle = 1000, lag = 5, plot = TRUE)

推定値を取り出してもとのシミュレーションデータと比較
# 推定値などを格納 res <- blockpfGaussianOpt(sim$data, particle = 1000, lag = 5, plot = FALSE) # 推定値を取り出す estimate <- apply(res$values, 2, mean) # 前に作っていたdata.frameに追加 df <- cbind(df, estimate) # プロット ggplot(data = df) + geom_line(mapping = aes(x = iteration, y = state), colour = "magenta") + geom_line(mapping = aes(x = iteration, y = estimate), colour = "green") + geom_point(mapping = aes(x = iteration, y = data), shape = 21, size = 3, colour = "blue")
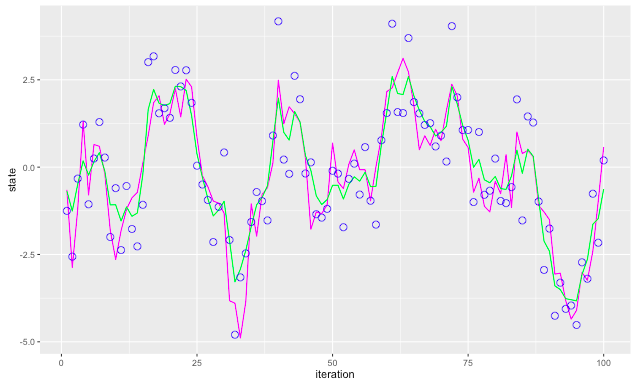
緑の線が推定値で、そこそこ推定できているとも言えなくもない結果がでてきました。
おわりに
他にもpfLineartBS、pfNonlinBSといった関数が用意されていますが、マニュアルに載っている論文の結果を再現するだけです。他のパッケージとしては、{SMC}もありますが、こちらはパッケージが2011年から更新されていないようです。こちらも後日時間があったらテストしてみたいと思っています。
Rでスパースモデリング:Group Lasso
はじめに
スパース推定の代表例として、Lasso(Least Absolute Shrinkage and Selection Operator)があります。
これは、正則化項としてノルムを加えるようなパラメータ推定法です。
このような正則化項を導入すると、大部分のパラメータは0に潰れてスパースな解が得られます。
それに加えて、近年、Lassoは様々な拡張モデルが提案されています。
その中の一つが、グループ化された変数がある場合に適用されるGroup Lassoというモデルです。
各グループを指定するラベルをとすると、Group Lassoの罰則項は以下のように書かれます。
ここで、各グループに関するノルムはではなく
ノルムです。
このように設定することにより、同じグループ内の変数がすべて残る/一斉に0になるという傾向をもつようになります。このあたりの直感的(図形的)説明は、Group Lassoでグループごと重みが0に潰れる理由 | Preferred Researchを参照してください。
Group Lassoの使いどころ
カテゴリカル変数そのものの効果をモデルに反映させたい
例えば、都道府県を説明変数に入れる場合があります。素朴には、各都道府県をフラグ化(and 適切にreferenceを設定)して、変数選択やLassoなどでどのような都道府県がモデルの変数として選択させる、などを調べることがあると思います。しかしながら、この方法だと、「都道府県じたい」が効果のある変数かどうかを判定したい場合にはうまく働きません。このようなあるカテゴリカル変数じたいの効果の有無を調べる場合にGroup Lassoは効果を発揮します。
先見的に似た変数の効果がわかっていて、それらを同時にモデルに反映させたい
遺伝子情報などの中には、あらかじめ効果がわかっているものがあることが少なくありません。特に、似たような効果をもつ遺伝子を同時にモデルに反映させたい場合に、Group Lassoが有効です。
Group Lassoの種類
Group Lassoには大雑把に重複なしGroup Lasso、重複ありGroup Lassoの2種類があります。前者は説明変数間で重複がないようにグループ化されている場合で、後者は説明変数間で重複を許すようにグループ化されている場合になります。この2種類は、どういった目的のモデルを作成したいかで使い分ける形になるでしょう。
RでGroup Lasso
Rに限っただけでもLassoの拡張モデルを扱えるパッケージはたくさんあります。ここでは、{grpreg}というパッケージを使ってGroup Lassoの挙動を見てみましょう。
{grpreg}のreferenceは以下になります。
https://cran.r-project.org/web/packages/grpreg/grpreg.pdf
パッケージをインストールし、サンプルデータ`Birthwt`を使用します。
if(!require(grpreg)){ install.packages("grpreg", dependencies = TRUE) }; require(grpreg) data(Birthwt)
ここで、Birthwtの中身はざっくり以下になっています。
- X : matrix 説明変数(妊婦の属性) 標準化の操作は済
- bwt : numeric 乳児の体重[kg] 線形回帰とかを想定した変数
- low : integer 低体重(2.5kgがthreshold)なら1, そうでなければ0 ロジスティック回帰とかを想定した変数
- group : factor あらかじめ変数を手でグループ分けしておいた結果を格納
説明変数の雰囲気を見ておきます。

それぞれの変数は以下です。
- age1~3 : 妊婦の年齢に関連する変数
- lwt1~3 : 妊婦の体重に関連する変数
- white, black : 妊婦の人種 その他(黄色人種とか)がreference
- smoke : 妊婦が喫煙歴あるか
- ptl1, ptl2m : 早産の回数フラグ 1回、2回以上
- ht : 高血圧の有無
- ut : 子宮過敏性の有無
- ftv1, 2, 3m : 妊娠の3ヶ月目までで医者を呼んだ回数フラグ 1回、2回、3回以上
Group Lassoで乳児の体重を妊婦の状態から説明するモデルを構築してみます。モデル構築には、grpreg関数を利用します。
model.grplasso <- grpreg(X = Exp, y = Obj, group, penalty="grLasso")
grpreg関数は、penaltyで罰則項の選択が可能です。grLassoはいわゆる普通のGroup Lassoで、他にはGroup SCAD, MCP, Bridgeなども選べる余地があります。さらに、{glnmet}のようにalphaをいじってElastic net的なモデリングも可能になっています。残差分布はGausian, Binomial, Poissonから選択できます。罰則の強さは何点かでグリッドサーチして求めており、基準はBIC (free energyの鞍点近似)がデフォルトになっています。ただし、自分で細かい調整がしたい場合はcv.grpregという関数を使うとより詳細なグリッドサーチが可能です。
実際に選択された変数を見てみましょう。
coef(model.grplasso)
(Intercept) 3.02538345542349 age1 0.12939932166821 age2 0.514120194706666 age3 0.307216803283638 lwt1 0.628666306376423 lwt2 -0.151307766326747 lwt3 0.49392658258674 white 0.167709378969912 black -0.0534652473427624 smoke -0.174393102956969 ptl1 -0.157879134519634 ptl2m 0.0444118454553915 ht -0.26668574037556 ui -0.36973322736348 ftv1 0 ftv2 0 ftv3m 0
確かに、グループ単位で0になる/ならないが表現されているモデルになっています。
おわりに
カテゴリカル変数の効果を見たい、先見的に似たような変数がわかっていてそれらの効果を取り込みたい、といった状況は実社会でもたびたびあると思われます。そのような場合は、Lassoだけでなく、Group Lassoを試してみると、新たな価値が生まれるかもしれません。
RでTopological Data Analysis : パッケージ(順次更新予定)
今回は、RでTopological Data Analysis(TDA)を行うためのパッケージとその概要を紹介します。
TDAのパッケージは、Rでもいくつか開発されています。
代表的なものは{TDA}、他にも{TDAmapper}、{phom}などあるようです。
本ブログでは、{TDA}を代表例として紹介していきます。
パッケージのマニュアルはこちら
- https://cran.r-project.org/web/packages/TDA/TDA.pdf : {TDA}
- https://cran.r-project.org/web/packages/TDAmapper/TDAmapper.pdf : {TDAmapper}
- http://www2.uaem.mx/r-mirror/web/packages/phom/phom.pdf : {phom}
{TDA}
{TDA}の関数
ざっくり以下の2種類
- データ点の間の距離を計算する関数
- 単純なEuclid距離から、Kernelで重みをつけた場合もある
- Persistence Diagramを計算する関数
- 元となるPersistent Homology群の構成法が複数あるため、それに応じて関数もいくつかある
{TDAmapper}
調査次第更新
{phom}
調査次第更新
Persistence DiagramやPersistent Homology群という見慣れない言葉が出現しましたが、これは次回以降、Rでデモをやりつつ紹介していきます。