Rで粒子フィルタ:RcppSMCのテスト
経緯
Rで粒子フィルタ(逐次モンテカルロ)ってできるんだっけ、とふと思ったので少し調べてみました。すると、Rのパッケージでは、例えば
- https://cran.r-project.org/web/packages/SMC/SMC.pdf : {SMC}
- https://cran.r-project.org/web/packages/RcppSMC/RcppSMC.pdf : {RcppSMC}
というパッケージがあるようです。{RcppSMC}の方は、Rcppで開発されているパッケージです。まずは、簡単にテストできそうな{RcppSMC}を使ってみることにしました。
参考記事
ネットで探しているときに目についた方のブログを参考にしました。
というかほぼ丸パクリです。
d.hatena.ne.jp
ito-hi.blog.so-net.ne.jp
また、粒子フィルタに関する書籍も載せておきます。自分が読んだことあるものは、例えば以下です。ざっくり概要を知るのは樋口さんの本のほうがよさげです。
www.kspub.co.jp
時系列解析入門https://www.iwanami.co.jp/.BOOKS/00/4/0054550.htmlwww.iwanami.co.jp
テスト
パッケージのインストール
if(!require(RcppSMC)){ install.packages("RcppSMC", dependencies = TRUE) } require(RcppSMC)
テストデータの生成
線形ガウス状態空間モデルのシミュレーションデータを生成します。データ点は特になんの思想もなく100点にしました。
n <- 100 sim <- simGaussian(len = n)
simはlistで、状態変数(state)と、観測データ(data)が格納されています。
プロット
無駄に{ggplot2}で描きます。
# パッケージのインストール if(!require(ggplot2)){ install.packages("ggplot2", dependencies = TRUE) } require(ggplot2) # data.frameに変換 df <- data.frame(iteration = 1:length(sim$state), state = sim$state, data = sim$data) # プロット ggplot(data = df) + geom_line(mapping = aes(x = iteration, y = state), colour = "magenta") + geom_point(mapping = aes(x = iteration, y = data), shape = 21, size = 3, colour = "blue")

観測データから状態を推定
ここでは、逆に与えられた観測データから状態変数を推定します。{RcppSMC}では、blockpfGaussianOpt関数を使用して行います。plot = TRUEとすると、自動的に図を出力してくれます。ここで、粒子数は1000としています。
blockpfGaussianOpt(sim$data, particle = 1000, lag = 5, plot = TRUE)

推定値を取り出してもとのシミュレーションデータと比較
# 推定値などを格納 res <- blockpfGaussianOpt(sim$data, particle = 1000, lag = 5, plot = FALSE) # 推定値を取り出す estimate <- apply(res$values, 2, mean) # 前に作っていたdata.frameに追加 df <- cbind(df, estimate) # プロット ggplot(data = df) + geom_line(mapping = aes(x = iteration, y = state), colour = "magenta") + geom_line(mapping = aes(x = iteration, y = estimate), colour = "green") + geom_point(mapping = aes(x = iteration, y = data), shape = 21, size = 3, colour = "blue")
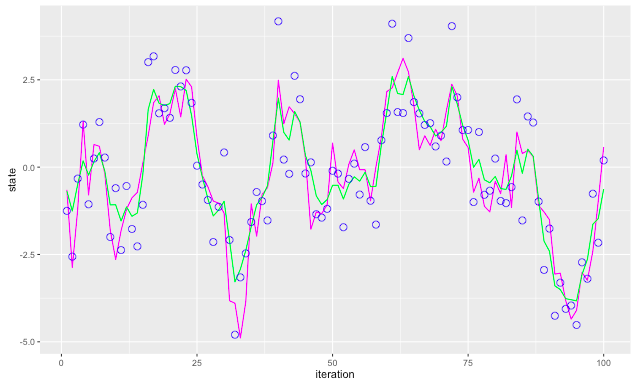
緑の線が推定値で、そこそこ推定できているとも言えなくもない結果がでてきました。
おわりに
他にもpfLineartBS、pfNonlinBSといった関数が用意されていますが、マニュアルに載っている論文の結果を再現するだけです。他のパッケージとしては、{SMC}もありますが、こちらはパッケージが2011年から更新されていないようです。こちらも後日時間があったらテストしてみたいと思っています。
Rでスパースモデリング:Group Lasso
はじめに
スパース推定の代表例として、Lasso(Least Absolute Shrinkage and Selection Operator)があります。
これは、正則化項としてノルムを加えるようなパラメータ推定法です。
このような正則化項を導入すると、大部分のパラメータは0に潰れてスパースな解が得られます。
それに加えて、近年、Lassoは様々な拡張モデルが提案されています。
その中の一つが、グループ化された変数がある場合に適用されるGroup Lassoというモデルです。
各グループを指定するラベルをとすると、Group Lassoの罰則項は以下のように書かれます。
ここで、各グループに関するノルムはではなく
ノルムです。
このように設定することにより、同じグループ内の変数がすべて残る/一斉に0になるという傾向をもつようになります。このあたりの直感的(図形的)説明は、Group Lassoでグループごと重みが0に潰れる理由 | Preferred Researchを参照してください。
Group Lassoの使いどころ
カテゴリカル変数そのものの効果をモデルに反映させたい
例えば、都道府県を説明変数に入れる場合があります。素朴には、各都道府県をフラグ化(and 適切にreferenceを設定)して、変数選択やLassoなどでどのような都道府県がモデルの変数として選択させる、などを調べることがあると思います。しかしながら、この方法だと、「都道府県じたい」が効果のある変数かどうかを判定したい場合にはうまく働きません。このようなあるカテゴリカル変数じたいの効果の有無を調べる場合にGroup Lassoは効果を発揮します。
先見的に似た変数の効果がわかっていて、それらを同時にモデルに反映させたい
遺伝子情報などの中には、あらかじめ効果がわかっているものがあることが少なくありません。特に、似たような効果をもつ遺伝子を同時にモデルに反映させたい場合に、Group Lassoが有効です。
Group Lassoの種類
Group Lassoには大雑把に重複なしGroup Lasso、重複ありGroup Lassoの2種類があります。前者は説明変数間で重複がないようにグループ化されている場合で、後者は説明変数間で重複を許すようにグループ化されている場合になります。この2種類は、どういった目的のモデルを作成したいかで使い分ける形になるでしょう。
RでGroup Lasso
Rに限っただけでもLassoの拡張モデルを扱えるパッケージはたくさんあります。ここでは、{grpreg}というパッケージを使ってGroup Lassoの挙動を見てみましょう。
{grpreg}のreferenceは以下になります。
https://cran.r-project.org/web/packages/grpreg/grpreg.pdf
パッケージをインストールし、サンプルデータ`Birthwt`を使用します。
if(!require(grpreg)){ install.packages("grpreg", dependencies = TRUE) }; require(grpreg) data(Birthwt)
ここで、Birthwtの中身はざっくり以下になっています。
- X : matrix 説明変数(妊婦の属性) 標準化の操作は済
- bwt : numeric 乳児の体重[kg] 線形回帰とかを想定した変数
- low : integer 低体重(2.5kgがthreshold)なら1, そうでなければ0 ロジスティック回帰とかを想定した変数
- group : factor あらかじめ変数を手でグループ分けしておいた結果を格納
説明変数の雰囲気を見ておきます。

それぞれの変数は以下です。
- age1~3 : 妊婦の年齢に関連する変数
- lwt1~3 : 妊婦の体重に関連する変数
- white, black : 妊婦の人種 その他(黄色人種とか)がreference
- smoke : 妊婦が喫煙歴あるか
- ptl1, ptl2m : 早産の回数フラグ 1回、2回以上
- ht : 高血圧の有無
- ut : 子宮過敏性の有無
- ftv1, 2, 3m : 妊娠の3ヶ月目までで医者を呼んだ回数フラグ 1回、2回、3回以上
Group Lassoで乳児の体重を妊婦の状態から説明するモデルを構築してみます。モデル構築には、grpreg関数を利用します。
model.grplasso <- grpreg(X = Exp, y = Obj, group, penalty="grLasso")
grpreg関数は、penaltyで罰則項の選択が可能です。grLassoはいわゆる普通のGroup Lassoで、他にはGroup SCAD, MCP, Bridgeなども選べる余地があります。さらに、{glnmet}のようにalphaをいじってElastic net的なモデリングも可能になっています。残差分布はGausian, Binomial, Poissonから選択できます。罰則の強さは何点かでグリッドサーチして求めており、基準はBIC (free energyの鞍点近似)がデフォルトになっています。ただし、自分で細かい調整がしたい場合はcv.grpregという関数を使うとより詳細なグリッドサーチが可能です。
実際に選択された変数を見てみましょう。
coef(model.grplasso)
(Intercept) 3.02538345542349 age1 0.12939932166821 age2 0.514120194706666 age3 0.307216803283638 lwt1 0.628666306376423 lwt2 -0.151307766326747 lwt3 0.49392658258674 white 0.167709378969912 black -0.0534652473427624 smoke -0.174393102956969 ptl1 -0.157879134519634 ptl2m 0.0444118454553915 ht -0.26668574037556 ui -0.36973322736348 ftv1 0 ftv2 0 ftv3m 0
確かに、グループ単位で0になる/ならないが表現されているモデルになっています。
おわりに
カテゴリカル変数の効果を見たい、先見的に似たような変数がわかっていてそれらの効果を取り込みたい、といった状況は実社会でもたびたびあると思われます。そのような場合は、Lassoだけでなく、Group Lassoを試してみると、新たな価値が生まれるかもしれません。
RでTopological Data Analysis : パッケージ(順次更新予定)
今回は、RでTopological Data Analysis(TDA)を行うためのパッケージとその概要を紹介します。
TDAのパッケージは、Rでもいくつか開発されています。
代表的なものは{TDA}、他にも{TDAmapper}、{phom}などあるようです。
本ブログでは、{TDA}を代表例として紹介していきます。
パッケージのマニュアルはこちら
- https://cran.r-project.org/web/packages/TDA/TDA.pdf : {TDA}
- https://cran.r-project.org/web/packages/TDAmapper/TDAmapper.pdf : {TDAmapper}
- http://www2.uaem.mx/r-mirror/web/packages/phom/phom.pdf : {phom}
{TDA}
{TDA}の関数
ざっくり以下の2種類
- データ点の間の距離を計算する関数
- 単純なEuclid距離から、Kernelで重みをつけた場合もある
- Persistence Diagramを計算する関数
- 元となるPersistent Homology群の構成法が複数あるため、それに応じて関数もいくつかある
{TDAmapper}
調査次第更新
{phom}
調査次第更新
Persistence DiagramやPersistent Homology群という見慣れない言葉が出現しましたが、これは次回以降、Rでデモをやりつつ紹介していきます。
Topological Data Analysis : 導入
ブログを作ったまま1年以上放置していました(笑)。 最近、アウトプットをしていこうという機運が高まっていているため、 ブログを積極的に活用していきたいと思います。
近年、Topological Data Analysisという、 位相幾何学をベースにしたデータ分析の手法が注目され始めています。 位相幾何学とは、通称「柔らかい幾何学」とも呼ばれており、 ざっくりいうと連続的な変形で移りあえる対象は「同じ」とみなすような学問です。
よくある例えとして、コーヒーカップとドーナツの例があります。
コーヒーカップが切れない粘土のような素材でできているとすると、
コーヒーカップを少しずつ(ちぎったりしないで)変形していくと、ドーナツに変形することができます。

このとき、コーヒーカップとドーナツの共通点は、「穴が一つ空いている」です。 これは非常にざっくりした特徴ですが、一方で雑多なものを取り払ったときに残る本質とも考えられます。
このような、位相幾何学のざっくりだが本質的な情報を取り出すことができる性質が、 データ分析の分野で注目され始めています。 これは、データの背後にある隠れた特徴が位相幾何学的な手法で抽出できるのではないか、と期待されているためです。 実際に、アメリカではAYASDIという位相的データ解析を専門としたベンチャー企業がすでに存在しており、 ベンチャーキャピタルから多額の融資を受けていることからも、その注目度の大きさが伺えます。 現在はまだDeep Learningのように一世を風靡する段階ではありませんが、 将来のブレイクスルー如何によっては大流行するかもしれません。
本ブログでは、Topological Data Analysisがどのようなものなのかを紹介していきたいと考えています。この分野に関しては、専門家でも何でもないので、何か間違ったことを公表してしまうかもしれません。その場合は、ぜひご意見いただけると嬉しいです。
ブログを始めてみた
はじめまして、tekenukoと申します。 今年の3月に素粒子理論で博士号を取得しました。 4月から民間企業でデータサイエンティスト(仮)として働きます。
大学院時代は主に超対称性を持つ素粒子模型の現象論を研究していました。 しかし、素粒子業界の現状を鑑み、より自分が面白いと思う分野へキャリアチェンジしたいと考えるようになりました。 特に、統計学や機械学習およびそれらの実社会での応用に興味を持ったため、データサイエンス業界に転じることにしました。
まだまだ基礎知識やスキルが不足している見習いですが、頑張って一人前のデータ分析屋になるために頑張ろうと思っています。 ここでは、自分の備忘録も兼ね、(仮)をとるためにいろいろ勉強したことのまとめなどを書いていきたいと考えています。 特に、理論の研究を専門にしてきたため、その経験を活かした内容になれば新しく誰かの役にたつのかな、と期待しています。
それでは、どうぞよろしくお願いいたします。